
Ilustración
1 RecA de E. Coli
Universidad Francisco Marroquín
Facultad de Medicina
Bioquímica Humana II
Fenómenos Genéticos
Roberto Viau
20000577
4 de octubre de 2001
Introducción
La generación de diversidad genética es muy importante para la adaptación de una especie. Al haber gran diversidad se permite que la especie se pueda adaptar a un mayor número de circunstancias. La diversidad se puede generar básicamente por dos mecanismos. El primero es la mutación al azar. Este mecanismo fue el primero en desarrollarse evolutivamente, pero tiene una gran desventaja: las probabilidades de obtener una mutación útil son muy bajas. Aunque toda diversidad se tuvo que haber iniciado de esta forma, se ideó un mecanismo para poder compartir este éxito del azar con otros miembros de la misma especie, y esta es la recombinación genética.
Recombinación
La recombinación genética consiste en tomar una secuencia de ADN de una fuente cualquiera e incorporarla a otra secuencia de ADN. El ejemplo más fácil es con bacterias: una bacteria puede tomar por conjugación un cromosoma de otra bacteria, o parte de él, tenerlo en forma de plásmido, e integrar el plásmido a su cromosoma central.
La recombinación está involucrada en una serie de reordenamientos genéticos. Aunque se ha estudiado principalmente en procariotes, parece que sus mecanismos son muy parecidos a los eucariotes inferiores. En mamíferos no se conoce el mecanismo exacto. (West, 1992) El mecanismo consiste en que una hebra de ADN invade a una hebra de ADN receptor. En seguida se forma un complejo heteroduplex, o complejo de Holliday. Finalmente, se resuelve el complejo cuando las dos hebras se separan, pero conteniendo información diferente.
Originalmente se identificó la proteína RecA en E. coli como la responsable de la recombinación. En humanos se ha encontrado la proteína HPP-1, que se cree puede tener un papel análogo a la RecA. (West, 1992)
La reacción catalizada por RecA se puede dividir en varios pasos. Primero, RecA se une a una cadena de ADN de una sola hebra, formando una fibra nucleoproteíca, En seguida, esta fibra se asocia con una hebra de ADN duplex de secuencia homóloga. Esta asociación se base en interacciones entre bases. Es posible sólo en los puntos donde las dos hebras tienen la misma secuencia. Esto limita la recombinación a secuencias muy parecidas. Estas tres hebras de ADN unidas forman una hélice. Es necesario que una de las hebras tenga una discontinuidad. Es a partir de esta, en una dirección 5' a 3', y a una velocidad de 2 a 10 pares de bases por segundo que se cambian los nucleótidos. Este es el caso más sencillo de recombinación, y a partir de un ADN linear duplex y uno circular de una hebra, se obtiene un ADN circular duplex y uno lineal de una sola hebra. Sin embargo la reacción más frecuente en eucariotes involucra dos secuencias duplex de ADN. Este caso es más interesante, porque adicionalmente el intercambio de información es recíproco, y no unidireccional como en el caso anterior. RecA no puede catalizar la formación del complejo de cuatro hebras directamente. Necesita que un corto segmento de una de las dos hebras duplex sea de una sola hebra. RecA se une a este segmento. Luego, como en el caso anterior, hay asociación de bases homólogas, y luego intercambio de secuencias, en la misma direccionalidad, pero un poco más lento. El intercambio se inicia en el segmento de una sola hebra,y esta debe tener entre 40 y 100 bases para alcanzar mayor eficiencia. (West, 1992)
La polaridad en estas reacciones sólo es importante en casos de secuencias circulares, ya que cuando son lineares, como en el caso eucariotas, carece de relevancia. En esta situación existe la polaridad, pero esta se define con respecto a cualquiera de las dos hebras.
El intercambio de fragmentos empieza en el fragmento complementario a la hebra más corta. (es decir, se inicia en el fragmento de sólo un haz de ADN) Todas las demás hebras tienen que ser homólogas al duplex del fragmento cortado. El producto final queda con fragmentos no unidos, y partes discontinuas, las cuales son reparadas por mecanismos convencionales. (West, 1992)
Todo este proceso conlleva cierto uso de ATP. RecA parece no necesitar ATP para catalizar ninguna de las reacciones antes descritas. Resulta que es poco afín al ADN. Debido a que el primer paso consiste en su unión a ADN, es necesario romper esta barrera. Para tal fin es que tiene una actividad de ATPasa. Sin embargo, en experimentos se ha suprimido esta actividad, y se ha logrado vencer la barrera aumentando el linking number del ADN, cambiando el pH, o usando Z-ADN. Se cree que el ATP funciona al enlazarse a RecA. En este momento se hidroliza, pero el ADP queda unido a la proteína. Este grupo funciona como efector alostérico, volviendo la molécula más afín al ADN. Al separarse RecA del ADN se libera el ADP. (West, 1992)
Una única RecA no basta para llevar a cabo todo el proceso. Se calcula que se una 1 RecA cada 3 pares de bases. El complejo helicoidal formado por RecA y los cuatro filamentos de ADN cuenta con 6 RecA y 18.6 pares de bases por vuelta.(West, 1992)

Ilustración
1 RecA de E. Coli
La unidad necesaria para la recombinación incluye 6 RecA. Esta proteína consta de dos regiones unidas por un segmento flexible (el segmento verde claro en la ilustración). Entre cada monómero existen interacciones proteína-proteína. La unión al ADN no es específica de secuencia, y ocurre en la hendidura menor, haciendo los contactos con el esqueleto de azúcar – fosfato.

Ilustración
2 Unión de RecA al ADN
Como se mencionó, RecA se asocia al ADN para forma una fibra nucleoproteíca. En el momento de intercambio de material, se cree existe una rotación del polímero de RecA, provocando que el un filamento de ADN duplex sea "enhebrado" al complejo. Una vez se logra esto se puede eliminar la RecA y queda una complejo de Holliday clásico. (West, 1992)
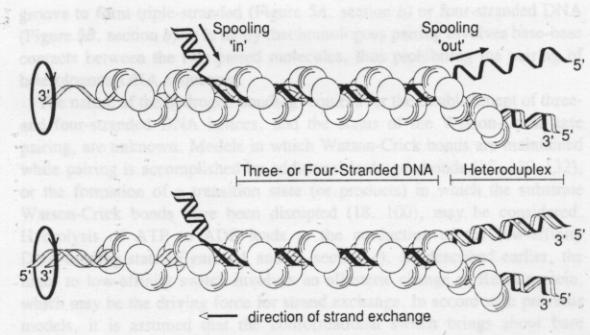
Ilustración
3 Enhebrando ADN
Ya se trataron los principales requerimientos para la recombinación, sin embargo, también están involucradas otras enzimas. Aquí se incluyen la ADN polimerasa, ligasa, topoisomerasa, y RecBCD. Estas enzimas se encontraron originalmente en procariotas, y es difícil pensar que tienen las mismas funciones en eucariotas, principalmente lo referente a RecBCD. RecBCD reconoce una secuencia Chi (GTCGGTGG), donde es más probable una inserción de ADN exógeno. (West, 1992)
En eucariotes surge el problema que todo el ADN es duplex, por lo que en teoría no se debiera dar recombinación, al no ser en los telómero. Sin embargo, en levaduras se ha detectado la actividad de una 3' exonucleasa. Esta actividad se encuentra en Sep1, el equivalente de RecA en levadura. Sep1 es una proteína más grande que RecA, por lo que se une una sola unidad por cada 12 pares de bases. También se han detectado muchas enzimas RAD que se piensa están involucradas con la reparación de rompimientos de los filamentos de ADN. RAD51 tiene ciertos dominios muy parecidos a RecA. (West, 1992)
Como se mencionó al principio, HPP-1 está involucrada con la recombinación en humanos. No se conoce mucho sobre ella. Se sabe que promueve el intercambio de hebras en una dirección 3' a 5', contrario al RecA. HPP-1 no tiene actividad de ATPasa, pero trabaja con otras enzimas que probablemente si la tienen. HPP-1 también posee actividad exonucleasa 3'-5'. Este último descubrimiento pon en duda la similitud en el mecanismo de recombinación usado por RecA y HPP-1. Es probable que HPP-1 se asocie primero a otras proteínas, y esto significaría un cambio en el mecanismo. (West,1992)
Sin importar el mecanismo, hasta ahora hay pruebas que todo termina en un complejo heteroduplex (de Holliday). Estos intermediarios deben de ser resueltos por rupturas endonucleolíticas. Existen dos posibles formas en que se puede resolver el heteroduplex, esto es un patch, o un splice. En el primero, una hebra de cada duplex queda intacta, mientras que en en splice, las dos sufren recombinación. La reacción mediante la cual se decide la resolución es muy compleja, e incluye varias rotaciones de 180 grados del ADN. Las rupturas del intermediario heteroduplex son catalizadas por distintas enzimas en procariotes que en eucariotes. En los segundos, existe una Endo X1 y X2. La primera actúa en secciones no homólogas, y produce un corte asimétrico, mientras que la otra lo hace en secuencias homólogas, y su corte es simétrico. Existen otras endonucleasas que participan en la resolución del complejo, y que cortan de maneras distintas. Muchas de ellas se han podido aislar de células HeLa humanas.
Reordenamiento Genético
Parte del proceso de generar diversidad genética exige que exista cierto orden. Incluso, muchas veces es posible lograr diversidad uniendo partes ya existentes. Es aquí donde entra el reordenamiento genético. Este consiste en una serie de procesos, la mayoría vinculados con la recombinación.
Recombinación selectiva
Se mencionó que uno de los principios elementales para la recombinación era que existiera cierta homología entre las secuencias. También se dijo que en eucariotes existían unas secuencias Chi, donde era más probable que ocurriera una recombinación. Una recombinación en este sitio sería una recombinación dirigida a sitio. (West, 1992; Nelson y Cox, 2000) Este rearreglo puede ocurrir en varias etapas de la célula. Puede estar involucrado en la regulación de la expresión de algún gen. También es el mecanismo que usan los fagos y algunos virus (retroviruses) para incorporarse al genoma de la célula huésped. La existencia de este mecanismo no se ha demostrado en células de eucariotes superiores, pero no existe razón para creer que no exista (West, 1992). Para que ocurra se necesita la acción de una recombinasa, como RecBCD para que junto a una enzima como RecA catalicen la formación del complejo heteroduplex, el cual se resuelve de manera convencional. La recombinasa puede tener acción de exonucleasa y de ligasa. (Nelson y Cox, 2000)
Este sistema puede ocurrir con segmentos dentro del mismo cromosoma. En este caso, un segmento se vuelve circular y se pierde (represión por deleción), mientras que otro segmento queda insertado en un nuevo sitio ya sea en forma convencional o inversa. (Nelson y Cox, 2000)
Conversión
Otro rearreglo genético muy relacionado es la conversión. Este término se suele usar laxamente para describir el fenómeno anterior. (Wysocki y Gefter, 1989) En un sentido más extricto, consiste en un intercambio no recíproco de información genética homóloga. Sería el caso en que el fragmento generado no se pierda, sino que quede incorporado en algún lugar del ADN. Esto significa que un gen se puede duplicar independientemente del genoma. Este fenómeno, por supuesto, es muy controlado, y se cree que con excepción de la generación de anticuerpos constituye un accidente. Este rearreglo puede ocurrir dentro de un mismo cromosoma, o puede ser entre dos cromosomas. Se cree que este fue el mecanismo mediante el cual se generaron diversas copias del gen de la globina fetal en mamíferos. También se cree que está involucrado con la duplicidad de los genes de histocompatibilidad (MHC I y II) mayor. (Wysocki y Gefter, 1989)
Transposición
Otro fenómeno de rearreglo es la transposición. Este consiste en "genes que saltan" de un lugar a otro. Hay quienes creen que son los precursores de los virus, algún tipo de gen parasítico. Su mecanismo de acción se compara mucho con la incorporación de virus y fagos al genoma central. Estos genes se insertan en un lugar más o menos aleatorio. Existen dos tipos principales: las secuencias de inserción, que sólo contienen la información necesaria para saltar y los transposones complejos, que acarrean más información. Los transposones tienen en sus orillas pequeñas secuencias repetidas. En el momento de incorporarse a un gen, se replican ciertas secuencias en el ADN receptor. Luego, se inserta el transposón. El resultado es que queda una hebra con un pedazo de más. Las enzimas de reparación se encargan de sintetizar la hebra complementaria. Cuando el transposón se copia para otro lugar, sucede lo mismo: se rompe en la secuencia repetida, y deja un espacio, el cual es regenerado por las enzimas de reparación. (Nelson y Cox, 2000) En un sistema más complejo, primero ocurre copia del transposón, y luego este se incorpora a otro lugar por medio de recombinación dirigida a sitio. Se forma un intermediario, el cointengrando, muy parecido al heteroduplex. Este es el mecanismo usado por los virus. (Nelson y Cox, 2000)
Durante el mecanismo de transposición, lo primero que sucede es que se exponen los extremos 3' del elemento transposón. Estas puntas son las que se enlazan con los extremos 5' del ADN receptor. Esto es lo más común, pero algunos elementos se insertan al inverso. La inserción inicial no está unida de un extremo, el cual debe ser reparado por una ligasa. Estas reacciones son catalizadas por integrasas u otras enzimas, muchas veces codificadas en el mismo transposón. (Craig, 1995)
Amplificación
Finalmente, hay otro rearreglo genético, la amplificación. Este puede o no estar relacionado con la recombinación. Una célula puede necesitar una gran cantidad de ARNm para cumplir sus necesidades de cierta proteína. La necesidad puede ser tan grande que sea necesario tener más de un gen para la misma proteína. En esto consiste la amplificación: en generar más copias de un gen. La amplificación puede ser permanente o temporal. Un ejemplo del primer caso serían las varias copias de globina fetal ya descritas. Durante el desarrollo, sin embargo, se puede dar el segundo caso. En esta situación, algún gen se duplica, y después de cierto número de divisiones celulares desaparece. Más común son las amplificaciones patológicas. Se cree que estas están involucradas en el cáncer. Influencian mecanismos como la capacidad de formar metástasis, y la resistencia a drogas. Debido a que la amplificación ocurre en gran modo por azar, las células que logran amplificar alguna resistencia a las drogas que suprimen la duplicación celular tienen una mayor ventaja de sobrevivir. Al final, del tumor desaparece todas las células susceptibles al medicamento y se reemplazan por células resistentes. (Stark y Wahl, 1984)
Físicamente, los genes amplificados se pueden encontrar en tres modalidades. Pueden ser fragmentos cromosómicos circulares, cromosomas lineales pequeños o áreas de un cromosoma con una tinción rara. Los primeros dos tipos suelen ser temporales, y eventualmente desaparecen después de un número variable de divisiones celulares. Las amplificaciones intracromosómicas son por su parte permanentes, y se conservan en las células hijas. (Stark y Wahl, 1984)
Los mecanismos mediante los cuales ocurre la amplificación son variados, incluso entre la misma especie. El primero ya fue descrito como conversión. Este produce amplificaciones intracromosómicas. En otro tipo de amplificación, ocurre replicación del genoma fuera del ciclo celular. La replicación excesiva de un punto selectivo causa un complejo mitóticamente inestable, que degenera a la formación de cromosomas circulares. También, en otro modelo, puede haber un error en la formación del cointegrando durante la transposición. El resultado es un cromosoma lineal extra. (Stark y Wahl, 1984; Wysocki y Gefter, 1989)

Ilustración
4 Mecanismo de amplificación por duplicación fuera de
tiempo
Consecuencias
Aunque ya se mencionaron las consecuencias principales de estos fenómenos, vale la pena volverlas a mencionar y hace énfasis en algunas. Como se dijo, el principal impacto de la recombinación es la generación de diversidad, de una forma menos arriesgada que la mutación al azar. Este es el mecanismo usado por los animales de reproducción sexual. Durante la meiosis, al formarse la tétrada, (Meiosis I, diplotene) ocurre recombinación entre los cromosomas homólogos. En esta recombinación se intercambian fragmentos de cromosomas y es la razón por lo que no siempre se sigue un patrón de herencia mendeliana.
La transposición / recombinación dirigida a sitio es muy importante para la infección viral. Muchos retroviruses son acusados de ser carcinógenos. Esto es porque al insertarse pueden activar el aparecimiento de cáncer. Al insertarse en un sitio pueden interrumpir algún gen que reprime la replicación incontrolada, o pueden activar uno que la provoque.
Durante la vida de la célula, los errores en el ADN pueden ser muy comunes. Generalmente se tratan de reparar por mecanismos como excisión de bases, de nucleótidos, reparación directa, etc. Sin embargo, cuando estos métodos ya no pueden usarse porque se ha perdido información, se usa una recombinación de emergencia. En este caso, se toma el cromosoma homólogo para sintetizar la parte del gen faltante. El mecanismo es muy parecido al de recombinación normal, sólo que no se producen los complejos heteroduplex, ya que el intercambio es sólo unidireccional.
La amplificación, como ya se mencionó, está relacionada al aparecimiento de resistencia por parte de células tumorales, además de estar involucrada en su aparecimiento en sí. También permite suplir una gran demanda de proteínas
Anticuerpos
Muchos de estos fenómenos están relacionados con la producción de anticuerpos y su diversidad. Los anticuerpos son un producto reciente de la evolución. Aparecieron súbitamente en la división de vertebrados con y sin mandíbula, quedando en los primeros. El sistema inmune que se desarrolló es tan complejo que puede producir una cantidad de anticuerpos mucho más grande de lo que se esperaría tomando en cuenta el tamaño de los genes que lo codifican. (Agrawal, 2000)
Un anticuerpo está formado por una cadena pesada y una ligera. Cada una de estas regiones tiene una región constante y otra variable. Las dos cadenas se unen por medio de una unión J. Cada uno de estos segmentos está codificado por un gen diferente. Sin embargo, existen varias versiones de cada uno de estos genes. De tal forma que se sugiere que una forma de generar la gran diversidad de anticuerpos es jugando con distintas combinaciones genéticas.(Agrawal, 2000)
Estas combinaciones se logran por medio de reacciones
muy parecidas a las transposiciones. De hecho, se cree que
evolucionaron de un transposón, el RAG. El procedimiento se
parece mucho al usado por el bacteriófago
![]() de
E. coli. El proceso inicia con un reconocimiento y rompimiento del
ADN entre el borde de una señal conservada de recombinación
y el segmento de interés. (recombinación dirigida a
sitio). Luego, se ligan las secuencias de recombinación para
formar una junta señal, se añaden o borran nucleótidos
antes de ligar para formar una junta de código. La primera
parte es mediada por proteínas RAG, y la segunda parte por las
enzimas normales de reparación de ADN. Las juntas de unión
requieren mucho procesamiento para incorporarse al ADN, sin embargo
lo hacen rápidamente. Por su parte, las juntas de señal
lo hacen más despacio. El resultado general de esto es que se
produzca una nueva secuencia de ADN, a partir de un banco de genes.
Los genes de donde salen estos fragmentos son pseudogenes, porque de
no ser por esta transposición no se expresarían. Los
pseudogenes entonces invaden un gen que si se transcribe, y forman un
composición al azar que permite que distintas células
puedan producir distintos anticuerpos. Este proceso ocurre solamente
en células en desarrollo. Se ha asociado su funcionamiento
anormal en casos de leucemia (Agrawal, 2000)
de
E. coli. El proceso inicia con un reconocimiento y rompimiento del
ADN entre el borde de una señal conservada de recombinación
y el segmento de interés. (recombinación dirigida a
sitio). Luego, se ligan las secuencias de recombinación para
formar una junta señal, se añaden o borran nucleótidos
antes de ligar para formar una junta de código. La primera
parte es mediada por proteínas RAG, y la segunda parte por las
enzimas normales de reparación de ADN. Las juntas de unión
requieren mucho procesamiento para incorporarse al ADN, sin embargo
lo hacen rápidamente. Por su parte, las juntas de señal
lo hacen más despacio. El resultado general de esto es que se
produzca una nueva secuencia de ADN, a partir de un banco de genes.
Los genes de donde salen estos fragmentos son pseudogenes, porque de
no ser por esta transposición no se expresarían. Los
pseudogenes entonces invaden un gen que si se transcribe, y forman un
composición al azar que permite que distintas células
puedan producir distintos anticuerpos. Este proceso ocurre solamente
en células en desarrollo. Se ha asociado su funcionamiento
anormal en casos de leucemia (Agrawal, 2000)
Sin embargo, las uniones entre cada gen no parecen hacerse fielmente. Se dice que sufren mutaciones somáticas, principalmente las regiones variables. Se podría creer que estas se deben a mutaciones puntuales, pero no parece ser probable. En cambio, se cree sucede una conversión genética entre las regiones V. La evidencia con respecto a esto viene principalmente del sistema inmune de las aves. Estas sólo tienen un gen V, por lo que no es posible justificar con el mecanismo anterior el aparecimiento de toda la gama de anticuerpos que producen. Estas conversiones, es decir cambios no recíprocos ocurren entre genes de cadenas cercanas. En caso de mamíferos, ocurre entre los mismos genes V. Para las aves, ocurre con los genes de otras cadenas. Estas conversiones son intracromosómicas y pueden causar un gran número de variaciones. (Wysocki y Gefter, 1989)
Las conversiones no sólo están involucradas en la variación durante el desarrollo del sistema inmune, sino que también en su evolución. Es factible pensar que la duplicidad de genes V en mamíferos se deba originalmente a este mecanismo. (Wysocki y Gefter, 1989)
Referencias
Wysocki, Lawrence; Gefter, Malcolm. 1989. Gene Conversion and the Generation of Antibody Diversity Ann. Rev. Biochem 58:509-31
Agrawal, Alka. 2000. Transposition and the Evolution of Antigen-Specific Immunity
Science 290 (Dec) 1715-1716
Craig, Nancy. 1995. Unity in Transposition Reactions
Science 270 (Oct) 253-254
Stark, George; Wahl, Geoffre.1989. Gene Amplification
Ann. Rev. Biochem 53:447-91
West, Stephen.1992. Enzymes and Molecular Mechanisms of Genetic Recombination
Ann. Rev. Biochem 61:603-40
Nelson, David; Cox, Micheal. 2000 Lehninger Principles of Biochemistry
3ed Edition. Worth Publishers
USA