Palindromati
fdocc arroba
yahoo punto com
Biotecnólogo Independiente.
RESUMEN
Este artículo describe a una familia de heterotrancritos
artificiales (quimeras de RNA), miles de secuencias en el Genbank que contienen fragmentos o al adaptador del tipo EcoRI ctcgtgccgaattcggcacgag actuando como enlazador palindrómico, el
cual une a dos o más genes producto de differentes cromosomas. Esto sucede
debido a que las metodologías actuales producen dichas secuencias que se
encuentran en el Genbank, así como en
los microarreglos de Affymetrix, y en
muchos artículos publicados que reportan o que usan dichas secuencias
portadoras del ligador del tipo EcoRI
dentro de sus regiones codficantes, y/o de sus sitios mRNA 5’UTR ó 3’UTRs. Este
ligando del tipo EcoRI y sus
heterotranscritos son considerados aquí como artefactos experimentales,
caracterización que pudiera ayudar a prevenir errores tanto en los estudios de
mecanismos moleculares así como en el descubrimiento de productos
farmacéuticos.
Palabras Clave: EcoRI, Palíndroma, Perilipina, Genbank, Affymetrix, microarreglo (microarray),
especies-específico.
INTRODUCCIÓN
Es vital en el descubrimiento de nuevos tratamientos
médicos el enfocarse en moléculas específicas sin efectos colaterales en otros
tejidos. Para lograr dicho objetivo es necesario un estricto control de calidad
dentro de las bases de datos moleculares. Este artículo describe el hallazgo de
numerosos artefactos metodológicos reportados al Genbank. Se recomienda el más detallado análisis de secuencias de
ácidos nucleicos para nuevos descubrimientos biológicos, médicos y
farmacéuticos.
Un único RNA atando en su sola hebra a dos
diferentes genes procedentes de dos diferentes cromosomas humanos (1) fue el principio
teorético para mi estudio de heterotranscritos.
Aquí defino heterotranscritos
como quimeras, secuencias compuestas de fragmentos correspondientes a dos o más
genes procedentes del uno o de dos o más cromosomas.
Consideré que dicho fenómeno reportado en
referencia (1) debería de ser reflejado en una racional y lógica combinación de
productos genéticos Inteligentemente Diseñados (2, 3). Ya que la mayoría de las
moléculas y de las rutas biológicas vitales se encuentran presentes en muchos
organismos, inicialmente pensé que el fenómeno descrito en la referencia (1)
tal vez estuviera presente en muchas secuencias naturales como posible común
denominador funcional.
Inicialmente supuse que el estudio de secuencias
similares o relacionadas con la presentada en la referencia (1), quizás pudiera
ser útil en nuestro entendimiento de las bases moleculares de la variabilidad
biológica.
Así, éste fenómeno se perfilaba como posible
explicación para la abundancia de proteínas que excede el número de sus genes,
debido a múltiples combinaciones modulares entre sus diversos mRNAs. Recientes
estimados para humanos consideran que existen más de un millón de proteínas
producidas por solamente unos 20,000 ó 25,000 genes (4).
Con dichas consideraciones en mente, mi idea
inicial fue que, si la referencia (1) estaba en lo correcto, entonces la
producción de numerosas proteínas podría haber experimentado un putativo
proceso de hetero-ensamblaje de RNA al momento de su formación.
Sin embargo, después de cinco años de comparar
secuencias, he llegado a percibir que esas hetero-secuencias que usan a un
mismo oligonucleótido como su ligador común, son únicamente artefactos
metodológicos.
El elemento común de éstas quimaeras es el ligador (el “linker”)
ccgaattcgg (que se presentó en referencia 1 dentro
de la secuencia L21934 para la enzima ACAT-1
del H. sapiens). Esto hace que las
referencias 1, 5 y 6 (si describieran a un fenómeno real), se refieran a un
evento especies-específico único para
los seres humanos (2, 3).
Otro artículo con la misma secuencia (1) ha sido
publicado recientemente por ese mismo grupo (5). Sus autores mencionaron desde
la referencia (1) la similitud de ese ligador con el adaptador mas uado de EcoRI (herramienta extensamente usada en
investigación en biología molecular), así de que la puerta sigue abierta para
verificar si éste es un artefacto metodológico o no (5).
La construcción inicial de esa secuencia demostró que su
librería de cDNA fue transformada en E.
coli (cepa MC1061) usando el
vector fagémido pBluescript, así como el vector de expresión pcDNA. Después, de nuevo lo retransformaron en la misma cepa de E. coli (6). Sin embargo, recientemente he visto que el uso de vectores similares pudiera
estar involucrado en la producción de artefactos quiméricos en múltiples
instancias, como en los ejemplos presentados en los Cuadros 1 y 2 (7).
Una posible, aunque remota, explicación para la
referencia (1) es que allí estamos tratando con un proceso natural,
exclusivamente restringido a los seres humanos. Así de que, cualesquiera que
sea el veredicto final, el hecho es que el ligador o adaptor del tipo EcoRI descrito en (1) fue el punto
inicial para los siguientes hallazgos que se describen en éste artículo.
Mi hipótesis personal es que los heterotranscritos o
quimeras que incluyen al ligador palindrómico del tipo EcoRI ctcgtgccgaattcggcacgag o
sus secuencias relacionadas, que se extienden al menos por unas doce bases, son
artefactos artificiales debidos a las metodologías moleculares usadas, evento
mediado principalmente por la interacción vector-hospedador.
RESULTADOS Y
DISCUSIÓN
El hallazgo de
un palíndroma similar en los microarreglos (microarrays) de Affymetrix
Las bases para este artículo aparecieron mientras
trabajaba con los microarreglos. Estaba yo estudiando los cambios en la
expresión genética en los ratones resistentes a la obesidad con la perilipina
inactivada, “knock-out” (8, 11),
mediante el uso de los DNA-Chips de Affymetrix,
MG-U74A-v2 y usando el software gratuito educacional dChip V.1.2 (9). Entonces un particularmente intrigante
hetero-transcrito se presentó, la secuencia nucleotídica AB030505, inicialmente
reportada por sus remitentes como si fuera el mRNA del Mus musculus para UBE-1c1, UBE-1c2 y UBE-1c3 (cds completos). A
continuación se describe la secuencia AB030505 y su elemento común, el ligador
del tipo EcoRI, presente en miles de
secuencias del Genbank.
Un cuidadoso estudio de la secuencia de nucleótído
AB030505 usando Blast (10) me dirigió
hacia un elemento que estaba ligando a dos largas secciones de dos diferentes
genes en un RNA:
1. La secuencia de nucleótido AK078792
del cromosoma 10, codificando para un homólogo de la proteína mutada ubicua de
melanoma (Mum1) y
2. La secuencia de nucleótido BC036273
del cromosoma 12, que codifica para la retinol deshidrogenasa 11 (similar al
Arsdr1).
El elemento de enlace dentro de la secuencia AB030505
resultó ser el palíndroma ctcgtgccgaattcggcacgag, compuesto
por 22 nucleótidos.
Aquí también, así como en el reporte inicial (1), dos
transcritos originados en dos diferentes cromosomas aparecieron pegados en una
sola hebra de mRNA. 22 bases centrales conteniendo al ligador, el palíndroma ccgaattcgg,
similar al inicialmente reportado por la referencia (1).
Una secuencia palindrómica para la doble hélice de DNA
posee los mismos nucleótidos si se lee en la dirección de 5’ a 3’, que es la
dirección normal de lectura, ya sea desde la hebra plus (+) o la hebra minus
(-). Una evaluación manual y visual de éste ligador palindrómico se llevó a
cabo. Sorprendentemente, éste ligador se encontró presente en miles de
secuencias reportadas al Genbank.
El Cuadro 2 expandido (7) presenta numerosos ejemplos del
palíndromo (o secuencias relacionadas) que han sido reportados por sus remitentes
como si se encontrara dentro de las regiones codificantes. El ligador
palindrómico es frecuentemente traducido como el péptido artificial RAEFGT, ausente en las bases de datos
de secuencias de proteínas (10).
Incremento en el número de secuencias
palindrómicas reportadas al Genbank
Un
incremento mensual ha sido observado en el número de secuencias que contienen
al ligador del tipo EcoRI o sus
derivados dentro de miles de secuencias. En un ejemplo reciente (14 Oct. 2005)
efectuado en Blastn (alineamiento nucleótido - nucleótido), seleccionando
la base de datos para las secuencias de ácidos nucleicos no redundantes (nr) del Genbank, una consulta de 44 letras
palindrómicas se usó:
CTCGTGCCGAATTCGGCACGAGCTCGTGCCGAATTCGGCACGAG
Con esta secuencia obtuve 6010 Blast Hits usando las siguientes condiciones:
1. 106 fue el número mínimo esperado. Algunos de
esos resultados se presentan en el Cuadro 2 (7).
2. 1000 fue el número de descripciones y de
alineamientos.
En
la base de datos alterna del Genbank
conteniendo secuencias incompletas expresadas (expressed sequence tags, est),
que son mRNAs de posibles proteínas, el número de secuencias conteniendo al
adaptador palindrómico EcoRI se
cuenta también en los miles.
Palíndromas adicionales encontrados
mediante el uso de microarreglos
Secuencias
adicionales pertenecientes a éstos ligandos también se encontraron mediante el
estudio de los resultados de microarreglos disponibles en el Internet usando la
mencionada herramienta de software dChip
(9) junto a la base de datos de las secuencias usadas por Affymetrix. En e1 Cuadro 1 se dan ejemplos conteniendo a los
ligadores palindrómicos.
Affymetrix ha sido una exitosa metodología de
microarreglos, para evaluar por ejemplo la expresión genética en humanos, en
ratones y ratas. Sin embargo, tanto la presencia de los heterotranscritos
artificiales y/o de sus ligadores artificiales pudiera conducir a una representación errónea de su expresión
genética dentro de los tejidos, ya que el área bajo la curva se reduce
dramáticamente para esas secuencias genéticas.
Cuadro 1. El ligador del tipo EcoRI se encuentra presente tanto en
secuencias del Genbank así como en
los microarreglos de Affymetrix para
humanos, ratones y ratas.
|
Clave/Organismo
|
EcoRI Affymetrix Sondas del Genbank [DNA Chip]
|
Gráfica con la
no expresión del ligador tipo EcoRI
|
Cita
|
|
AB002533_at
Homo sapiens
|
Gaattcggcacgagcacgcgtgaga,
Ggcacgagcacgcgtgagacttctc
[en los DNA Chips Humanos HuGene-FL y Hu6800]
|

Proteína Ribosomal LP2 (Qip1)
|
Shipp et al, Nature Med.
8, 68. 2002
|
|
AJ243503
(99534_at)
Mus musculus
|
Ttcggcacgagctcgtgccggtcct
[en los DNA Chips del Ratón MG-U74Av2 y MG-U74A]
|

Adipocito
Ghrelina
|
Castro-Chavez et al. Diabetes 52, 2666. 2003
|
|
AI045710
(rc_AI045710_at)
Rattus
norvegicus
|
Atgatatgtacagatccctcgtgcc,
tgatatgtacagatccctcgtgccg,
tatgtacagatccctcgtgccgcct,
tgtacagatccctcgtgccgcctcg,
gtacagatccctcgtgccgcctcgt
[en el DNA Chip de la Rata RG-U34B}
|
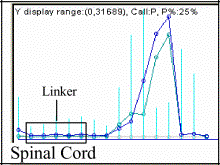
Disúlfido isomerasa, prot. relacionada
(Erp70)
|
Children's National
Medical Center
Visitado en Feb. 1, 2005.
|
Nota: El palindróma ctcgtgccgaattcggcacgag
del tipo EcoRI causa la caída en la expresión
genética registrada por el microarray,
demostrando su ausencia en los organismos [dChip
V.1.2 (9)]. En la seguna columna se resaltan en azul claro las porciones
correspondientes al linker palindrómico;
en azul obscuro, los nucleótidos que son reemplazados para obtener el segundo
set de sondas en "mismatch"
para los DNA-Chips de Affymetrix.
El fenómeno de
heterotranscripción
Doce bases parecen ser el mínimo común denominador para
que el ligando palindrómico EcoRI sea
capaz de producir heterotranscritos artificiales como los aquí reportados
presentes en el Genbank.
Los flancos palindrómicos más comunes para el oligo ccgaattcgg
son g y c, produciendo el
oligonucleótido gccgaattcggc. Menos frecuentes son los flancos c y g para producir el segundo
oligonucleótido cccgaattcggg, que posee un efecto similar de heterotranscripción.
Ésta última secuencia palindrómica es la que tenemos en la referencia (1). La
misma secuencia palindrómica se encuentra en el ejemplo 9 del Cuadro 2 (Homo sapiens X93499 para la proteína
RAB7), en la que nos encontramos con fragmentos de más de dos genes unidos en
la misma hebra, mediante los ligadores palindrómicos ccccgaattcgggg y gcccgaattcgggc (12).
Cuadro 2. RNAs con porciones del ligador
relacionado con EcoRI y algunas de
sus citas.
# |
Clave/Organismo |
Gene/Proteína * |
Linker ** |
Citas *** |
|
1
|
U58090
Homo sapiens |
Miembro de la familia de genes Hs-cul-4A |
aattcggcacgagctcgtgccgct
NSARARAA
|
Cell 85,
829-839. 1996.
|
|
2
|
U28831
Homo sapiens
|
Proteína
inmuno-reactiva con anticuerpos policlonales anti-PTH
|
gcacgagctcgtgccgat
ARARAD
|
Proc. Assoc. Am. Physicians 107, 296-305. 1995.
|
|
3
|
BC041619
Homo sapiens |
Proteína KIAA0404, para IMAGE:5923662 [R: Prot.
Hipotética MGC16044]
|
ccctcgtgccgaattcggcacgag
PSCRIRHE |
Proc. Natl. Acad. Sci. U.S.A. 99, 16899-16903. 2002. |
|
4
|
AF176705 Homo sapiens |
Proteína F-box FBX10 (PINX1) [R: vector] |
cctcgtgccgaattc PRAEF |
Curr. Biol. 9, 1180-2. 1999. |
5 |
X85792
Homo sapiens
|
Vpr, proteína de enlace 1
|
tcgtgccgaattcggcacgag
SCRIRHE
|
Benichou
et al. [Unpublished]; J Biol Chem. 277, 45091-8. 2002
|
6 |
AF151109
Homo sapiens
|
Proteína Putativa BRCA1-interactiva (BRIP1)
|
ggcacgagctcgtgccgc
GTSSCR
|
Wang et al. BRCA1-interacting protein.
[Unpublished]; Oncogene 19, 6152-8.
2000.
|
|
7
|
AF146697
Homo sapiens
|
FOXP1
|
aagaattcggcacgagct
KNSARA
|
Cancer Res. 61, 8820-8829. 2001.
|
|
8
|
NM_002342
Homo sapiens
|
Gene
y 3' UTR de la superfamilia
TNFR, miembro 3 (LTBR)
|
gctcgtgccgaattc
|
Genomics 16, 214-218. 1993. [Curated by NCBI]
|
|
9
|
X93499
Homo sapiens
|
Proteína RAB7, de enlace a GTP [L: Distroglicano 1. C: Rab7.
R: Glicoproteína de la cubierta]
|
ccccgaattcgggg
& gcccgaattcgggc
|
Biochem. Biophys. Res.
Commun. 229, 887-890. 1996.
|
|
10
|
X82200
Homo sapiens
|
Gene y mRNA de Staf50 inducido por interferón
|
gaattcggcacgagctc
|
J. Biol. Chem. 270, 14891-14898. 1995.
|
|
11
|
U31384
Homo sapiens
|
mRNA
para la Proteína G, subunidad g-11
|
ggcacgagctcgtgccg
|
J. Biol. Chem. 270, 21765-21771. 1995.
|
|
12
|
AF379619
Homo sapiens
|
Intrón de AB13, precursor del mRNA
|
gaattcggcacgagct
|
van Roy
and Staes. New human gene family. [Unpublished].
|
|
13
|
AY245868
Homo sapiens
|
CDS
del pseudogene de la Proteína del tipo
Aldehído oxidasa (AOX2)
|
aagaattcggcacgagca
LNSARA
|
Wright
RM. Human aldehyde oxidase. [Unpublished].
|
|
14
|
AF339764
Homo sapiens
|
mRNA del hígado y del bazo fetal IMAGE:108721
|
gaattcggcacgagcggcacgagct
|
Genomics 79, 635-656. 2002.
|
|
15
|
U43527
Homo sapiens
|
5'UTR
del supresor de metástasis del melanoma maligno KiSS-1
|
(ctct)15cctcgtgccgaattcggcacgag
(como aparece
en la secuencia reportada al Genbank)
|
J. Natl. Cancer Inst. 88, 1731-1737. 1996; Genomics
54, 145-148. 1998.
|
Notas: Aquí se presenta al ligador del tipo EcoRI ctcgtgccgaattcggcacgag como aparece en el Genbank
para algunos genes humanos. Para ver el resto de este Cuadro 2 y la presencia del linker en otros organismos, acuda al
URL: http://www.oocities.org/plin9k/t2.htm
* Gene/Proteína (Símbolo del Gene) [notas para los
flancos del linker (L or R)]
** Linker
y su Traducción a Amino Ácidos según el Genbank
*** Referencias Correspondientes de
acuerdo con el Genbank.
Abundancia de
secuencias que incluyen el linker palindrómico
del tipo EcoRI
Existen miles de secuencias que incluyen a las secuencias
incompletas expresadas (expressed
sequence tags, est) en el Genbank y en otras bases de datos de
nucleótidos que aún contienen artefactos artificiales que presentan como común
denominador a los linkers
palindrómicos EcoRI semejantes a
aquellos que aquí se describen.
Los linkers
palindrómicos pudieran presentarse en tándems, en mitades, o con diferentes
longitudes; siendo de 12 a 24 bases el rango más común. Los linkers artificiales se han reportado
aún dentro de múltiples regiones codificantes, tales como los ejemplos que se
presentan en el Cuadro 2 expandido (7). Éstos linkers también se reportan frecuentemente fuera de la región
codificante, como por ejemplo en sus promotores:
1-) NR_001557 del H. sapiens aldehído oxidasa 2 (AOH2) en el cromosoma 2, oligo gaattcggcacgagc
(13).
2-) NM_002342 H. sapiens receptor beta de linfotoxina (LTBR; miembro 3 de la
superfamilia TNFR), oligo gctcgtgccgaattc (14).
Además, dichos linkers
palindrómicos también han sido encontrados en la región 5’, v.gr., en la secuencia U43527 del
supresor de metástasis del melanoma maligno KiSS-1, oligo (ctct)15cctcgtgccgaattcggcacgag
(15), y/o en la región 3’, v.gr., en
la secuencia AY029161 para la proteína X1 que
interactúa con Pin2, oligo ctcgtgccgaattcggcac (16).
De los pocos envíos al Genbank que explícitamente reportan la presencia del adaptador EcoRI, Hirama et al (17) se destaca, junto con Inoue et al (18) y Savas et al
(19). Sin embargo, Inoue et al (18) consideran
solamente a las 8 bases iniciales del flanco izquierdo como si fueran parte del
adaptor EcoRI. El efecto del linker palindrómico para la secuencia de
Inoue pudiera extenderse al menos por unas 16 bases (secuencia D83948 para la
proteína S1-1, oligo ggcacgagctcgtgccg) mediante un
aparente fenómeno de auto-recombinación y auto-inserción en las interacciones
hospedero-vector.
Una situación similar a aquella que observamos en Inoue
se presenta en Savas et al (19),
quien menciona en su envío al Genbank
únicamente a las primeras 6 bases como si fueran parte del adaptor EcoRI (que pasa igual con la referencia
1, pero no con su secuencia enviada, L21934). En la referencia (19) de Savas et al, el efecto del tipo linker pudiera extenderse por unas 20
bases (secuencia X78445 para el citocromo P450 Cyp1-b-1, oligo gaattcggcacgaactcgtgc).
Hirama et al
(17) es el único que menciona una extensión mayor para el adaptor de EcoRI, reportando que ésta se extiende
por 14 bases (secuencia X56703 para la cadena rearreglada alfa del receptor de
células T, oligo gaattcggcacgagct).
Quimeras ligadas
por el palíndromo tipo EcoRI parecen
resistir a su digestión enzimática
Los linkers
palindrómicos persisten en las secuencias sin haber sido digeridos por sus
enzimas. El descubrimiento de los mecanismos precisos de resistencia a la
digestión enzimática se encuentra en espera de adicionales estudios. Sin
embargo, es evidente que los más comunes linkers
palindrómicos corresponden a la secuencia del adaptor de EcoRI gaattcggcacgag, que se usa para el sitio 5’UTR, y que ha sido
reportada en algunas instancias al Genbank,
v.gr., dentro de la secuencia
AI607511, ctcgtgccgaattcggcacgag (similar a fosfoproteína acídica
ribosomal PO). En dicha secuencia, se indica el uso del vector pBluescript SK(-), así como de EcoRI, con el uso adicional del vector Uni-ZAP XR. Dichas metodologías, así
como aquellas descritas en (1, 5, 6), pudieran promover el fenómeno de
ligamiento artificial abundante en el Genbank.
El rearreglo y escisión (splicing) en la interacción hospedero-vector resistente a la
digestión enzimática de EcoRI pudiera
explicarse como un fenómeno de auto-hibridación del linker (Figura 3), lo que pudiera hacerlo aparecer como si fuera un
apéndice imposible de ser capturado por la enzima digestiva.
Figura 3. La auto-hibridización del
ligador palindrómico del tipo EcoRI
pareciera bloquear la digestión enzimática. Fenómeno también observado como si
fuera un 'cierre de pantalón' a ambos
extremos de largas secuencias, en donde dos partes distantes del ligador se
aproximan y se pegan produciendo una forma tipo plásmido. [Ejemplos 38-42,
Cuadro 2 expandido (7). Ésta figura se obtuvo usando el software de la referencia 20.]
CONCLUSIONES Y
PERSPECTIVAS
Un control de calidad más
estricto en las bases de datos de secuencias
Éstos hallazgos pudieran contribuir a la implementación
de un más estricto control de calidad dentro de las bases de datos de nucleótidos,
así como el desarrollo de analistas profesionales de artefactos moleculares y
experimentos relacionados, aparte de la ingeniería racional de fármacos y
proteínas. Primeramente hemos de domar, mediante su uso controlado, a dichos linkers palindrómicos. Entonces, el uso
del control de calidad de secuencias (para detectar artefactos moleculares)
pudiera ser exitoso. Actualizaciones mejoradas pudieran llevarse a cabo en el
diseño de los microarreglos de Affymetrix,
v.gr., mediante la remoción del linker palindrómico de secuencias
humanas tales como AA557228, AA113291, AI798671, AA864645, AA780435, W90032,
AA810599, T52176, AA976510, AI380906, AA535275, AI792166, T67559, L04270,
U83598, D59474, T03148, T54342, D59674, D59787, D80164, H90908, N80129, C14426,
D59619, D80240, T56800, C14298, W72424, IR1056496, T69555, D80337, C14227,
C14407, C14344, D80210, etcétera.
Posible uso de palíndromas y
heterotranscritos para sanar enfermedades hereditarias
Otra posibilidad es el uso de palíndromas para el ensamblaje
de dos genes diferentes (módulos genéticos) para la ingeniería de proteínas
terapéuticas, v.gr., para el
desarrollo de tratamientos antiobesidad que pudieran ser personalizados y
prescritos de acuerdo con el metabolismo particular de cada persona (21).
Vectores artificiales para
la terapia génica pudieran producir heterotranscripción
Es posible que linkers
palindrómicos semejantes a los que se reportan en éste artículo, u otros linkers, pudieran ser producidos por
vectores artificiales usados en terapia genética. Si éste es el caso, pudiera
esperarse un efecto colateral inadvertido en humanos. A la fecha, éstos linkers siguen presentes en miles de
secuencias artificiales que son mandadas al Genbank
cual alegóricas de cosas que pudieran prevenirse en la terapia génica humana.
Éste artículo se ha escrito para alertar a la comunidad
científica de la presencia de éstos miles de hetero-transcritos artificiales
debidos a metodologías actuales que producen dichas secuencias reportadas al Genbank, así como el chequeo de los
vectores usados en terapia génica.
Heterotranscritos publicados y
enviados al Genbank siguen en espera
de su corrección
Analizando las secuencias presentadas como mRNAs
quiméricos en Tabla 4, referencia (22), encontré el linker palindrómico relacionado con EcoR1 dentro de la secuencia AY029161 (16), previamente mostrada.
En dicha secuencia se encuentra un fragmento para el LPTL, putativo supresor de
tumor humano, AF418553, originado en el cromosoma 8, ligado a un fragmento para
la fosfohistidina fosfatasa PHP14, NM_014172, originado en el cromosoma humano
9.
Hasta el momento de publicar este artículo, ninguna de
las secuencias aquí presentadas ha sido corregida en el Genbank. ¿Pudiera ésto deberse a que hasta la fecha no se ha determinado
que dichas secuencias son productos artificiales?
Otra secuencia en referencia (22) conteniendo al linker palindrómico EcoR1 era la secuencia BC000519, ya identificada como artefacto y
finalmente removida del Genbank.
La posible
remoción del linker palindrómico
semejante al transposón parece llevarse a cabo en otras quimeras
Esperando su corrección se tiene a miles de secuencias
semejantes a las que se presentan en el Cuadro 2 (7). Secuencias tales como la
AF176705 para la proteína F-box FBX10 humana (23), y la Z28355 del atrium de corazón (24), conteniendo
ambas fragmentos del vector y del linker.
Otra secuencia, HTCBYB08, contiene únicamente fragmentos del vector, careciendo
del linker. Experimentos se
realizaron también con dichas secuencias que carecen de evidencia alguna del linker palindrómico sin obtener
productos amplificados mediante RT-PCRs (resultados
no publicados). Ambas secuencias Z28355 y HTCBYB08 exhiben trazas del mismo
vector de clonación, vector de la secuencia X52324 (ARBLSKM), que es el vector pBluescript SK(-)(25-27). Una
auto-remoción del tipo transposón del linker
palindrómico pudiera ser una forma en la que muchos heterotranscritos carecen
de linker alguno, como en la
secuencia HTCBYB08. Ésta hipótesis también necesita ser experimentalmente
evaluada. Si éste resultara ser el caso, los linkers palindrómicos pudieran ser usados para la posible reversión
artificial de mutaciones in vivo
(28).
Por otro lado, heterotranscritos carentes del linker y presentes en los microarreglos
de Affymetrix pudieran mostrar dos
diferentes perfiles de expresión, por ejemplo, una expresión nula por uno de
sus lados, con una contrastante alta expresión por el otro lado (datos no mostrados). Un patrón similar
se presenta cuando los intrones no-expresados se incluyen en sondas de Affymetrix lado a lado con las
secuencias expresadas por los exones, como se ve en la Figura 1 de la
referencia (2).
Comentario Final
El palíndroma clave de éste artículo se ha presentado en
referencia (2) CTCGTGCCGAATTCGGCACGAG
y ha sido reportado en otros trabajos (2, 3, 11, 29).
AGRADECIMIENTO
Tracy Lynn Duncan ayudó a preparar esta revisión y me
soportó durante los últimos tres años.
REFERENCIAS
1. Li
BL, Li XL, Duan ZJ, Lee O, Lin S, Ma ZM, Chang CC, Yang XY, Park JP, Mohandas
TK, Noll W, Chan L & Chang TY (1999) Human Acyl-CoA:Cholesterol
Acyltransferase-1 (ACAT-1) gene organization and evidence that the 4.3-kilobase
ACAT-1 mRNA is produced from two different chromosomes. J. Biol. Chem. 274: 11060-11071.
2. Castro-Chavez
F (2004) Microarrays, antiobesity and the liver. Ann. Hepatol. 3: 137-145. [http://www.medigraphic.com/pdfs/hepato/ah-2004/ah044c.pdf]. Accessed Oct. 1, 2005.
3.
Castro-Chavez F (2004) Some Implications for the Study of Intelligent Design
Derived from Molecular and Microarray Analysis. PCID 3.1.7 (serial online), November, Issue 3. [http://www.iscid.org/pcid/2004/3/1/pcid_contents_2004_3_1.php].
Accessed Oct. 1, 2005.
4.
International Human Genome Sequencing Consortium (2004) Finishing the
euchromatic sequence of the human genome. Nature
431: 931-45.
5. Yang
L, Lee O, Chen J, Chen J, Chang CC, Zhou P, Wang ZZ, Ma HH, Sha HF, Feng JX,
Wang Y, Yang XY, Wang L, Dong R, Ornvold K, Li BL & Chang TY (2004) Human acyl-coenzyme
A:cholesterol acyltransferase 1 (acat1) sequences located in two different
chromosomes (7 and 1) are required to produce a novel ACAT1 isoenzyme with
additional sequence at the N terminus. J.
Biol. Chem. 279: 46253-62.
6. Chang
CC, Huh HY, Cadigan KM & Chang TY (1993) Molecular cloning and functional
expression of human acyl-coenzyme A:cholesterol acyltransferase cDNA in mutant
Chinese hamster ovary cells. J. Biol.
Chem. 268: 20747-55.
7. Castro-Chavez
F (2005) Full version of Table 2. Website. [http://www.oocities.org/plin9k/t2.htm].
Accessed Oct. 1, 2005.
8.
Castro-Chavez F, Yechoor VK, Saha P, Martinez-Botas J, Wooten E, Sharma S,
O’Connell P, Taegtmeyer H & Chan L (2003) Coordinated upregulation of
oxidative pathways and downregulation of lipid biosynthesis underlie obesity
resistance in Perilipin knockout mice. A microarray gene expression profile. Diabetes 52: 2666-2674. [http://diabetes.diabetesjournals.org/cgi/content/full/52/11/2666].
Accessed Oct. 1, 2005.
9. Li C & Wong WH (2001) Model-based analysis of oligonucleotide arrays: expression index computation and outlier
detection. Proc. Natl. Acad. Sci. U.S.A. 98: 31-36. [http://biosun1.harvard.edu/complab/dchip]. Accessed Oct. 1, 2005.
10. Altschul SF, Madden TL, Schäffer AA, Zhang J, Zhang Z, Miller W & Lipman DJ (1997) Gapped BLAST and
PSI-BLAST: A new generation of protein database search programs. Nucleic Acids Res. 25: 3389-3402
[http://www.ncbi.nlm.nih.gov/BLAST]. Accessed Oct. 1, 2005.
11. Castro-Chavez F (2004) El Incremento Coordinado de las Rutas Metabólicas Oxidativas y la Disminución en la
Biosíntesis de Lípidos Caracterizan la Resistencia a la Obesidad en el Ratón Artificialmente Carente de Perilipina.
Doctoral thesis, Universidad de Guadalajara, Guadalajara, Jalisco, Mexico.
[http://www.oocities.org/plin9k/tesis-fdocc.zip]. Accessed Oct. 1, 2005.
12. Vitelli R, Chiariello M, Lattero D, Bruni CB & Bucci C (1996) Molecular cloning and expression analysis of the human
Rab7 GTP-ase complementary deoxyribonucleic acid. Biochem. Biophys. Res. Commun. 229: 887-890.
13.
Terao M, Kurosaki M, Marini M, Vanoni MA, Saltini G, Bonetto V, Bastone A, Federico
C, Saccone S, Fanelli R, Salmona M & Garattini E (2001) Purification of the
aldehyde oxidase homolog 1 (AOH1) protein and cloning of the AOH1 and aldehyde
oxidase homolog 2 (AOH2) genes. Identification of a novel molybdo-flavoprotein
gene cluster on mouse chromosome 1. J.
Biol. Chem. 276: 46347-63.
14. Matsuda A, Suzuki Y, Honda G, Muramatsu S, Matsuzaki O, Nagano Y, Doi T, Shimotohno K, Harada T, Nishida E,
Hayashi H & Sugano S (2003) Large-scale identification and characterization of human genes that activate NF-kappaB and
MAPK signaling pathways. Oncogene 22: 3307-3318.
15. West
A, Vojta PJ, Welch DR. & Weissman BE (1998) Chromosome localization and
genomic structure of the KiSS-1 metastasis suppressor gene (KISS1). Genomics 54: 145- 148.
16. Zhou XZ & Lu
KP (2001) The Pin2/TRF1-interacting protein PinX1 is a potent telomerase
inhibitor. Cell 107: 347-359.
17.
Hirama T, Takeshita S, Matsubayashi Y, Iwashiro M, Masuda T, Kuribayashi K,
Yoshida Y & Yamagishi H (1991) Conserved V(D)J junctional sequence of
cross-reactive cytotoxic T cell receptor idiotype and the effect of a single
amino acid substitution. Eur. J. Immunol.
21: 483-488.
18.
Inoue A, Takahashi KP, Kimura M, Watanabe T & Morisawa S (1996) Molecular
cloning of a RNA binding protein, S1-1. Nucleic
Acids Res. 24: 2990-7.
19.
Savas U, Bhattacharyya KK, Christou M, Alexander DL & Jefcoate CR (1994)
Mouse cytochrome P-450EF, representative of a new 1B subfamily of cytochrome
P-450s. Cloning, sequence determination, and tissue expression. J Biol Chem. 269: 14905-11.
20. Zuker
M. (2003) Mfold web server for
nucleic acid folding and hybridization prediction. Nucleic Acids Res. 31: 3406-15.
21. Qi L, Shen H,
Larson I, Schaefer EJ, Greenberg AS, Tregouet DA, Corella D & Ordovas JM.
(2004) Gender-specific association of a perilipin gene haplotype with obesity
risk in a white population. Obes. Res.
12: 1758-65.
22.
Romani A, Guerra E, Trerotola M & Alberti S (2003) Detection and analysis
of spliced chimeric mRNAs in sequence databanks. Nucleic Acids Res. 31, e17: 1-8.
23.
Winston JT, Koepp DM, Zhu C, Elledge SJ & Harper JW (1999) A family of
mammalian F-box proteins. Curr. Biol.
9: 1180-1182.
24.
Auffray C, Behar G, Bois F, Bouchier C, Da Silva C, Devignes MD, Duprat S,
Houlgatte R, Jumeau MN, Lamy B (1995) IMAGE:
molecular integration of the analysis of the human genome and its expression. C. R. Acad. Sci. III, Sci. Vie. 318:
263-72.
25.
Short JM, Fernandez JM, Sorge JA & Huse WD (1988) Lambda ZAP: a bacteriophage lambda expression vector with in vivo excision properties. Nucleic Acids Res. 16: 7583-7600.
26.
Alting-Mees MA & Short JM (1989) pBluescript
II: gene mapping vectors. Nucleic
Acids Res. 17: 9494.
27. Alting-Mees MA, Sorge JA & Short
JM (1992) pBluescriptII:
multifunctional cloning and mapping vectors. Meth. Enzymol. 216: 483-495.
28.
Hirschhorn R (2003) In vivo reversion
to normal of inherited mutations in humans. J.
Med. Genet. 40: 721-728. [http://jmg.bmjjournals.com/cgi/content/full/40/10/721]
Accessed Oct. 1, 2005.
29. Castro-Chavez F (2005) Genes involucrados en la
susceptibilidad a la obesidad y los genes anti-obesidad. In: Mendez-Sanchez
N (Editor), OBESIDAD. Epidemiología,
Fisiopatología y Manifestaciones Clínicas. 2nd Ed. Manual
Moderno. Mexico (in press).
////////////////////////////////////////////////////////////////
Notas:
El PDF de éste
trabajo en inglés ‘Palindromati’ se puede descargar de:
http://www.iscid.org/boards/ubb-get_topic-f-6-t-000582.html
El HTM de éste
trabajo en inglés ‘Palindromati’: http://www.oocities.org/plin9k
Bioingeniería Mendeliana:
http://www.oocities.org/plin9k/bioeng.htm
Blog en inglés
“Research on Intelligent Design”
(Investigación en Diseño Inteligente):
Tasters of the Word (YouTube), videos recientes: "Astronomía y Nacimiento de Jesucristo: Once de Septiembre Año Tres A.C.", "Estudio sobre Sanidades" (en 20 episodios), "Jesus Christ, Son or God?":http://www.youtube.com/1fertra
Tasters of the Word (the blog, with: "Astronomy and the Birth of Jesus Christ"):http://fertra1.blogspot.com



