MOLECULAR MODELLING AND STRUCTURAL ANALYSIS OF PAPAIN

Papain compared to Arabidopsis thaliana proteins |
||||||||||||||||||||||||
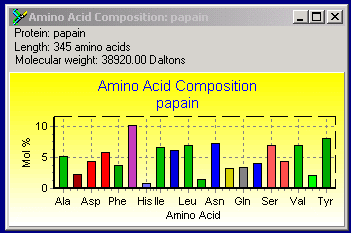
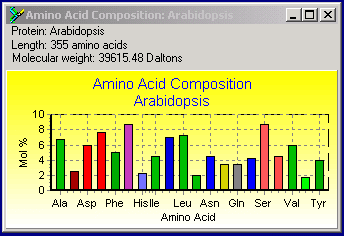
Graphical representation of the amino acid composition of Papain and Cyteine Preteinase of Arabidopsis thaliana. ( 1) |
||||||||||||||||||||||||
SEARCH RESULT OF A MATCH OF PAPAIN AGAINST THE
ARABIDOPSIS THALIANA PROTEIN
|
||||||||||||||||||||||||
|
Protein from different sources
and sometimes diverse biological functions can have similar sequences,
and it is generally accepted that high sequence similarity is reflected
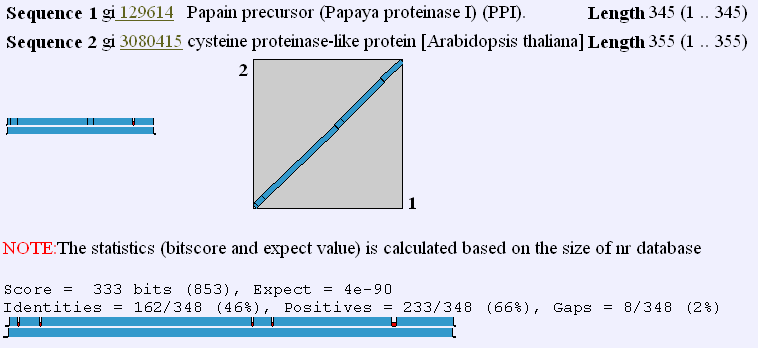
by distinct structure similarity. A FASTA search of the Papain precursor
(P00784)against the Arabidopsis thalian proteins yielded the following results:-
./align/31625.3662109375A: 345 aa
>Your Sequence; vs ./align/myThal-descr.seq library The best score obtained from the search
searching ./align/myThal-descr.seq library opt E() < 20 1 0:= 22 0 0: one = represents 39 library sequences 24 0 0: 26 2 1:* 28 1 6:* 30 12 35:* 32 105 135:===* 34 377 367:=========* 36 778 754:===================* 38 1308 1247:===============================*== 40 1726 1739:============================================* 42 2110 2126:======================================================* 44 2240 2345:========================================================== * 46 2335 2388:===========================================================* 48 2275 2286:==========================================================* 50 2140 2086:=====================================================*= 52 1815 1834:===============================================* 54 1557 1567:========================================* 56 1276 1309:=================================* 58 1066 1074:===========================* 60 908 870:======================*= 62 723 698:=================*= 64 618 555:==============*= 66 455 439:===========* 68 347 345:========* 70 272 270:======* 72 228 211:=====* 74 181 165:====* 76 118 128:===* 78 102 100:==* 80 65 77:=* 82 71 59:=* 84 55 47:=* 86 30 36:* 88 21 28:* inset = represents 1 library sequences 90 27 22:* 92 24 17:* :================*======= 94 17 13:* :============*==== 96 10 10:* :=========* 98 8 8:* :=======* 100 6 6:* :=====* 102 5 5:* :====* 104 0 4:* : * 106 1 3:* := * 108 3 2:* :=*= 110 1 2:* :=* 112 1 1:* :* 114 1 1:* :* 116 1 1:* :* 118 1 1:* :* >120 34 0:= *================================== 11049032 residues in 25458 sequences Expectation fit: rho(ln(x))= 5.1207+/-0.000988; mu= 7.8428+/- 0.058; mean_var=78.0098+/-17.977 Kolmogorov-Smirnov statistic: 0.0069 (N=29) at 58. |
||||||||||||||||||||||||
Fig. 1 |
||||||||||||||||||||||||
|
|
||||||||||||||||||||||||
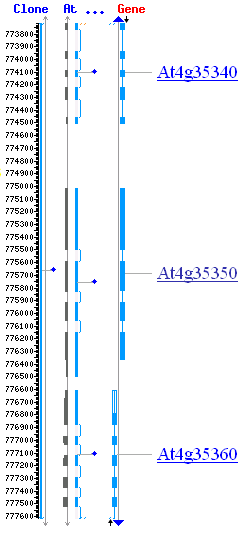
A. thaliana - FST matches for entry At4g35350(4) 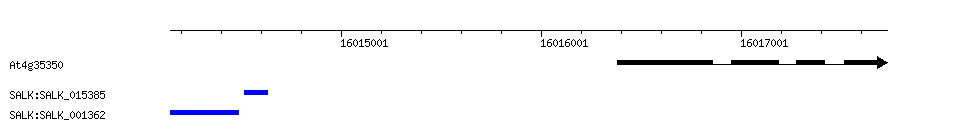
entry code entry coordinates At4g35350 >W/16016385-16016859,16016954-16017189,16017279-16017419,16017516-16017731 FST code match coordinates SALK:SALK_015385 W/16014520-16014635 SALK:SALK_001362 W/16014147-16014487 Legend Black gene model (coding region) Blue FST matches Boxes exons Horizontal lines introns |
||||||||||||||||||||||||
|
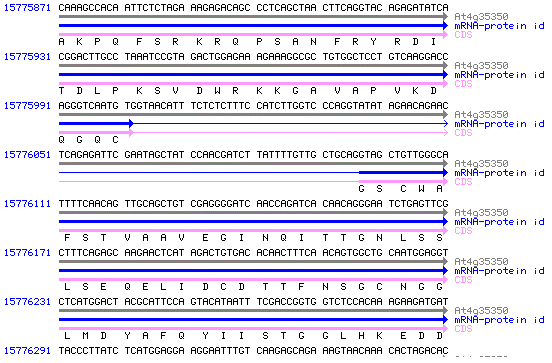
A. thaliana - mRNA matches for entry At4g35350
(contig dt_d_23)((5)
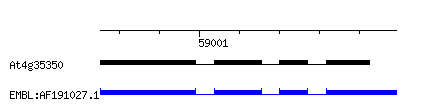
entry code entry coordinates At4g35350 W/58505-58979,59074-59309,59399-59539,59636-59851 mRNA code match coordinates on contig dt_d_23 EMBL:AF191027.1 W/58505-58979,59074-59309,59399-59539,59636-59984 Legend Black gene model (coding region) Blue mRNA matches (including UTRs) Boxes exons (4) Horizontal lines introns Vertical lines mRNA coordinate matches gene model coordinate |
||||||||||||||||||||||||
|
PREVIOUS
NEXT |